Nucle - PowerPoint PPT Presentation
1 / 79
Title: Nucle
1
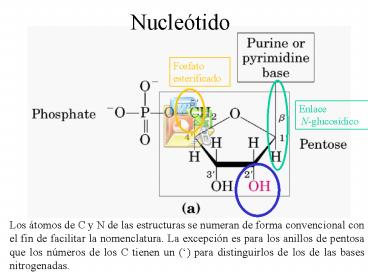
Nucleótido
Fosfato esterificado
Enlace N-glucosídico
Los átomos de C y N de las estructuras se numeran
de forma convencional con el fin de facilitar la
nomenclatura. La excepción es para los anillos de
pentosa que los números de los C tienen un ()
para distinguirlos de los de las bases
nitrogenadas.
2
Bases nitrogenadas
3
Bases nitrogenadas
1
9
1
4
Pentosa Ribosa
Libre, en disolución
RNA b-D-ribofuranosa DNA b-2-desoxi-D-ribofuran
osa
5
(No Transcript)
6
Desoxiribonucleótidos
7
Ribonucleótidos
8
Las células contienen también nucleótidos con
grupos fosfatos en posiciones diferentes del C
5.
Ribonucleósido 2,3-fosfato Cíclico
intermedios. Ribonucleósido 3-fosfato Son
Productos finales de hidrólisis del RNA.
9
Degradación del RNA en medio básico. Diferencia
con DNA.
10
Unión covalente de unidades de nucleótidos
consecutivas.
A pH 7.0 están cargados negativamente Ácido.
11
Extremos 5 y 3 diferenciados.
1
3
5
OLIGONUCLEÓTIDO Ácido nucleico de cadena corta.
50 o menos nucleótidos. POLINUCLEÓTIDOS De mayor
longitud.
12
Propiedades de las bases de los
nucleótidos influyen en la estructura de los
ácidos nucleicos.
Purinas y pirimidinas Débilmente básicas. Muchos
átomos del anillo participan de la resonancia y
por eso la mayor parte de los enlaces tienen
carácter parcial de doble enlace. Pirimidinas
planas. Purinas Con ligera torsión. Existen en
dos o más formas tautoméricas en función del
pH. En consecuencia de la resonancia absorben luz
UV (propiedad de Cada una). Hidrofóbicas a pH
neutros. Interacción hidrofóbica Apilamiento.
Interacciones de Van der Waals y de tipo
dipolo-dipolo. Enlaces puente de H. entre las
bases (pares específicos).
13
Formas tautoméricas del URACILO.
14
(No Transcript)
15
Doble Hélice.
16
ESTRUCTURA PRIMARIA Estructura covalente y su
secuencia de nucleótidos. ESTRUCTURA SECUNDARIA
Cualquier estructura regular y estable adoptada
por algunos o todos los nucleótidos. ESTRUCTURA
TERCIARIA Plegamiento complejo de grandes
cromosomas.
17
Miescher nucleína (núcleos de leucocitos). Aisló
DNA. DNA Herencia-1944-Avery, MacLeod y McCarty.
Streptococcus pneumoniae neumococo.
18
(No Transcript)
19
(No Transcript)
20
(No Transcript)
21
(No Transcript)
22
E. coli
23
Leyes de Chargaff 1- Composición de bases del
DNA varía de una especie a otra. 2- DNA aislado
de muestras de tejidos diferentes de la misma
especie tienen igual composición de bases. 3-
Composición de bases del DNA de una especie dada
no varía con el estado nutricional, ni con la
edad, ni con variaciones am- bientales. 4-
Independientemente de la especie, todo DNA tiene
AT y CG, entonces purinaspirimidinas
(AGTC).
24
Modelo de difracción de rayos x del DNA.
25
3,6 nm 10,5 bases por vuelta.
Eje longitudinal. Dos periodicidades. Doble
hélice dextrógira. Parte hidrofílica
externa. Parte hidrofóbica interna.
Modelo lineal Esqueleto covalente y apilamiento
de bases.
Modelo espacial lleno.
26
Diferente composición y secuencia de las
cadenas. Complementarias.
Puentes de hidrógeno Especificidad. Fuerzas de
Van der Waals Estabilidad.
27
Lógica de la REPLICACIÓN.
28
DNA molécula extraordinariamente flexible.
Capacidad de rotación alrededor de sus enlaces.
29
(No Transcript)
30
Desviaciones de la estructura de Watson y Crick.
Fluctuaciones térmicas La estructura se
curva, se estira o las bases se desaparean.
31
(No Transcript)
32
Ciertas secuencias Estructuras DNA no habituales.
poliA Torción del DNA Importancia en la unión
de proteínas al DNA.
Relación con funciones localmente importantes en
el metabolismo del DNA.
33
Palindromo pueden formar.
HORQUILLA
34
Palindromo pueden formar.
CRUZ
35
(No Transcript)
36
(No Transcript)
37
(No Transcript)
38
(No Transcript)
39
(No Transcript)
40
(No Transcript)
41
RNAm
42
Exón
Intrón no codificante
43
(No Transcript)
44
Típico empaquetamiento dextrógiro en una
hebra Simple de RNA.
45
Tipos de estructura secundaria observados en
algunos RNA.
46
Doble hélice en una horquilla.
47
(No Transcript)
48
Fenilalanina-tRNA de levadura.
49
Algunos apareamientos de bases no habituales.
50
Ribozima
51
Intrón Ribozima.
52
Desnaturalización del DNA por cambios de pH o
aumento de la temperatura.
La transición de la doble hebra de DNA a la mono
hebra desnaturalizada puede observarse por
disminución en la viscosidad o por aumento en la
Absorción de luz UV.
53
Tm punto de fusión.
54
Relación entre tm y contenido de C G
55
Micrografía de DNA parcialmente desnaturalizado.
56
Ensayo de hibridación.
A mayor relación evolutiva entre las especies,
mayor grado de hibridación de sus DNA.
57
Transformaciones no enzimáticas MUTACIONES.
Alteraciones espontáneas de su estructura
covalente.
58
(No Transcript)
59
Formación de dímeros de la timina por la luz UV.
60
(No Transcript)
61
Acelera la desaminación de bases.
62
Alteran ciertas bases del DNA.
63
Secuenciación del DNA por el método de Sanger.
64
(No Transcript)
65
(No Transcript)
66
(No Transcript)
67
(No Transcript)
68
(No Transcript)
69
(No Transcript)
70
(No Transcript)
71
(No Transcript)
72
(No Transcript)
73
(No Transcript)
74
Portadores de energía.
75
Coenzimas y cofactores enzimáticos.
76
(No Transcript)
77
(No Transcript)
78
(No Transcript)
79
Segundos mensajeros.